빅데이터와 인공지능(머신 러닝, 딥러닝)
- 세월이 흐르면서 쌓인 방대한 데이터를 빅데이터라고 함
- 이 ‘빅데이터’는 분명히 머신 러닝과 딥러닝으로 하여금 사람에 버금가는 판단과 지능을 가질 수 있게끔 했음.
빅데이터의 질
- 데이터 양도 중요하지만 그 안에 ‘필요한’ 정보가 얼마나 있는가가 중요
- 데이터 양이 많다고 무조건 좋은 결과를 얻을 수 있는 것은 아님
- 준비한 데이터가 사용하려는 머신 러닝과 딥러닝에 효율적으로 사용될 수 있도록 잘 가공되어 있는지 역시 중요
머신 러닝 프로젝트의 성공과 실패
- 얼마나 좋은 데이터를 가지고 시작하느냐에 영향을 많이 받음
- 여기서 좋은 데이터란?
- 한쪽에 치우지지 않고 불필요한 정보가 적으며 왜곡되지 않은 데이터를 의미
- 데이터를 잘 정제된 형식으로 변경이 필요
실습) 피마 인디언 데이터 분석
비만은 유전일까? 아니면 식습관 조절에 실패한 자신의 탓일까?

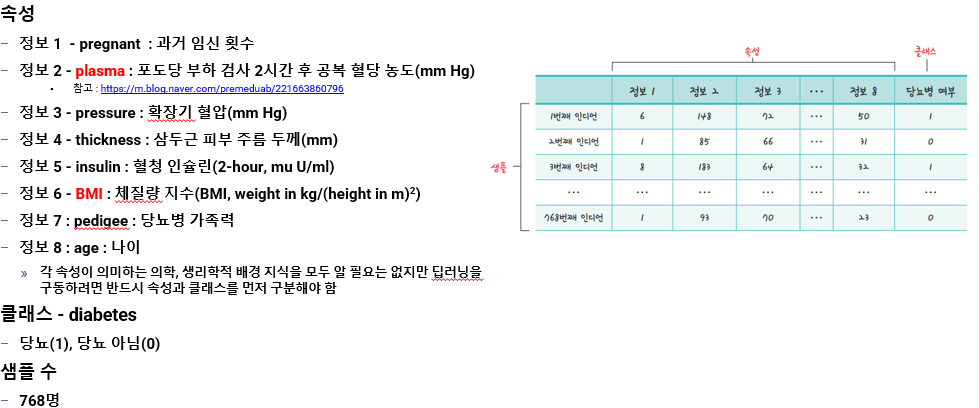
데이터 준비 및 확인
# 1. 환경 설정 - 모듈 import
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
# 2. 데이터 준비 - 깃허브에 준비된 데이터를 가져옵니다.
!git clone <https://github.com/taehojo/data.git>
# 피마 인디언 당뇨병 데이터셋을 불러옵니다.
df = pd.read_csv('./data/pima-indians-diabetes3.csv')
df.head(5)
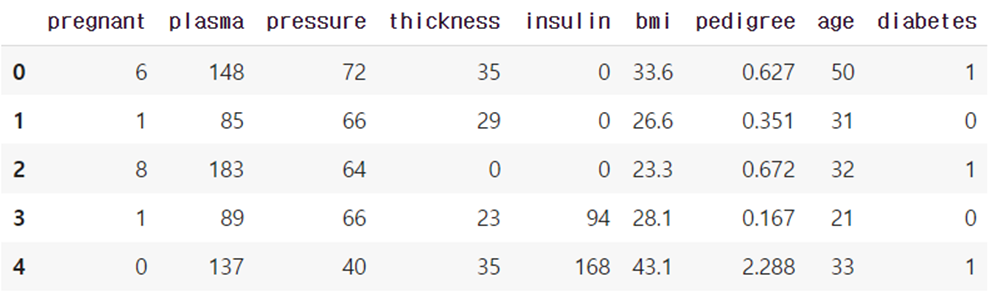
# 전체 데이터 크기를 확인
df.shape # ( 768, 9 )
# 1 : 당뇨병, 0 : 당뇨병 아님
df["diabetes"].value_counts()
# diabetes - 정상 500/ 당뇨병 268
# 0 500
# 1 238
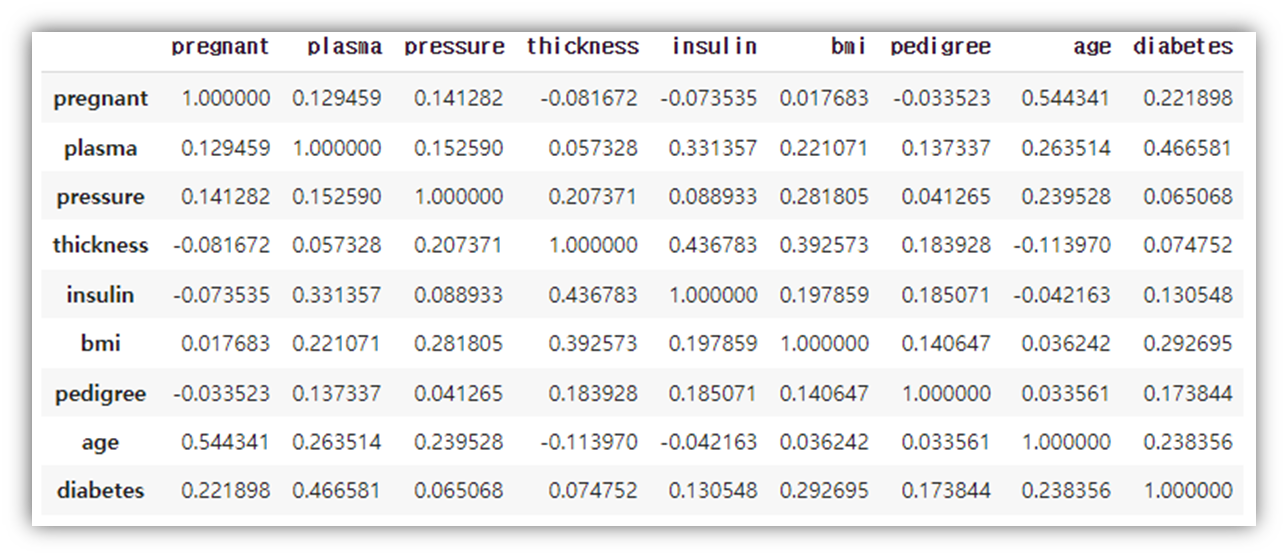
# 각 항목이 어느 정도의 상관 관계를 가지고 있는지 알아봅니다.
df.corr() # 상관계수(correlation) 출력
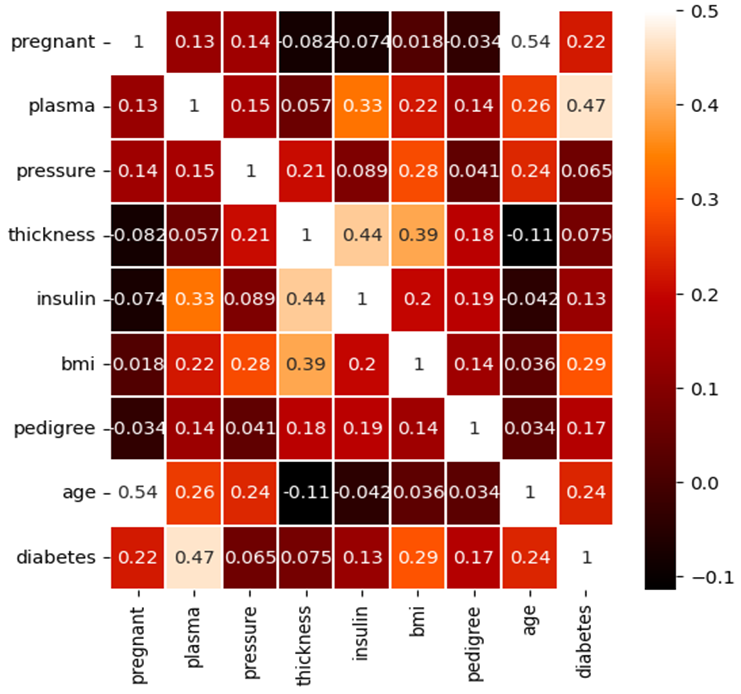
상관 관계 히트맵
# 데이터 간의 상관 관계를 그래프로 표현
colormap = plt.cm.gist_heat # 그래프의 색상 구성을 설정
plt.figure(figsize=(6,6)) # 그래프의 크기 설정
# 그래프의 속성을 결정. vmax의 값을 0.5로 지정해 0.5에 가까울수록 밝은색으로 표시sns.heatmap(df.corr(),linewidths=0.1,vmax=0.5, cmap=colormap,
linecolor='white', annot=True)
plt.show()
plasma 기준 정상과 당뇨 비율 확인
# plasma를 기준으로 각각 정상과 당뇨가 어느 정도 비율로 분포하는지 조사
plt.hist(x=[df.plasma[df.diabetes==0], df.plasma[df.diabetes==1]], bins=30,
histtype='barstacked', label=['normal','diabetes'])
plt.legend()
plt.xlabel('plasma')
plt.ylabel('number of sample')
plt.show()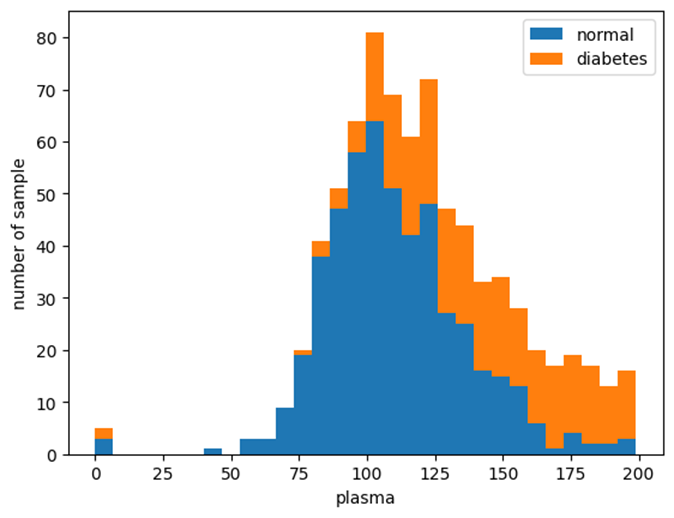
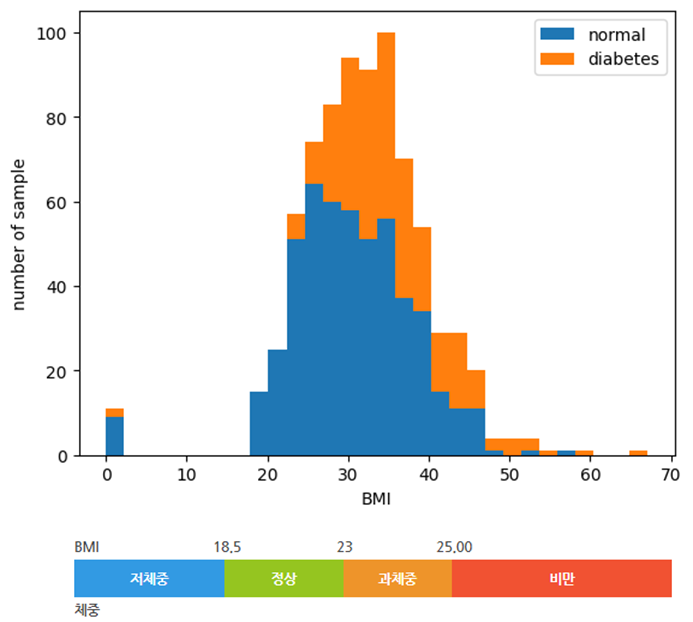
BMI 기준 정상과 당뇨 비율 확인
# BMI를 기준으로 각각 정상과 당뇨가 어느 정도 비율로 분포하는지 조사
plt.hist(x=[df.bmi[df.diabetes==0], df.bmi[df.diabetes==1]], bins=30,
histtype='barstacked', label=['normal','diabetes'])
plt.legend()
plt.xlabel('BMI')
plt.ylabel('number of sample')
plt.show()
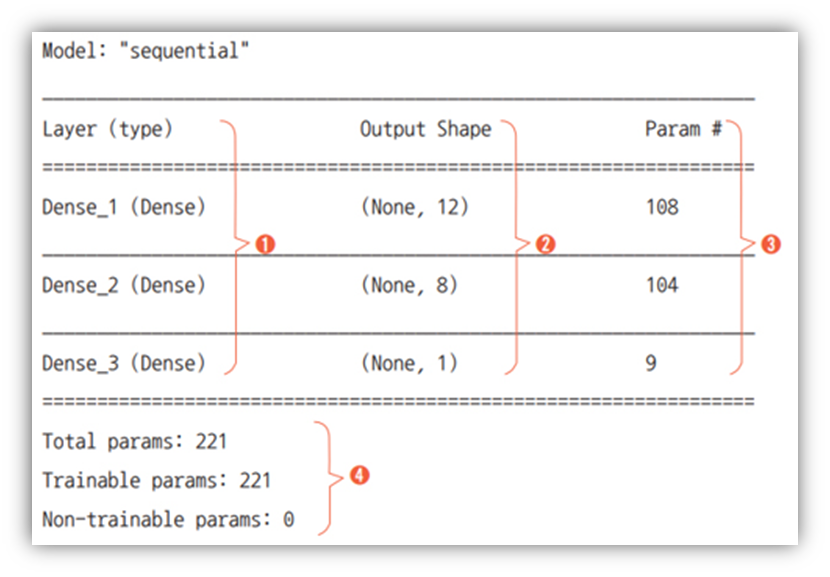
모델 구성
# 1.환경 설정
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
import pandas as pd
# 2. 데이터 준비
#!git clone <https://github.com/taehojo/data.git>
df = pd.read_csv('./data/pima-indians-diabetes3.csv’)
X = df.iloc[:,0:8] # 속성을 X에 할당
y = df.iloc[:,8] # 클래스(타겟)을 y에 할당
# 3. 모델을 구조 설정
model = Sequential()
model.add(Dense(12, input_dim=8, activation='relu', name='Dense_1'))
model.add(Dense(8, activation='relu', name='Dense_2'))
model.add(Dense(1, activation='sigmoid',name='Dense_3'))
model.summary()


# 4. 모델을 컴파일 및 실행
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['accuracy'])history=model.fit(X, y, epochs=100, batch_size=5)
약 71.61%의 정확도를 보이고 있음
상관도가 낮은 2개를 제외한 나머지 속성으로 예측 모델 생성
# 2. 데이터 준비
df = pd.read_csv('./data/pima-indians-diabetes3.csv’)
# pressure, thickness은 당뇨병 발병과 상관계수가 낮음.
df = df.drop(df.columns[2:4], axis=1)
# 세부 정보를 X로 지정합니다.
X = df.iloc[:,0:6]
# 당뇨병 여부를 y로 지정합니다.
y = df.iloc[:,6]
# 모델을 설정합니다.
model = Sequential()
model.add(Dense(12, input_dim=6, activation='relu', name='Dense_1'))
model.add(Dense(8, activation='relu', name='Dense_2'))
model.add(Dense(1, activation='sigmoid',name='Dense_3'))
#model.summary()
# 모델을 컴파일합니다.
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['accuracy’])
# 모델을 실행합니다.
history=model.fit(X, y, epochs=100, batch_size=6)

# 3. 모델을 구조 설정
model = Sequential()
model.add(Dense(14, input_dim=8, activation='relu', name='Dense_1'))
model.add(Dense(24, activation='relu', name='Dense_2'))
model.add(Dense(1, activation='sigmoid',name='Dense_3'))
# 모델을 컴파일합니다.
model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['accuracy'])
# 모델을 실행합니다.
history=model.fit(X, y, epochs=100, batch_size=6)
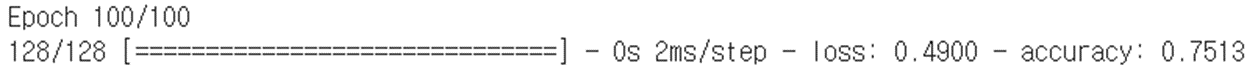
'빅데이터 분석가 양성과정 > Python - 딥러닝' 카테고리의 다른 글
| 모델 성능 검증 (0) | 2024.07.17 |
|---|---|
| 다중 분류 - iris 데이터 (0) | 2024.07.17 |
| 오류역전파 (0) | 2024.07.17 |
| 퍼셉트론(XOR) (0) | 2024.07.17 |
| 로지스틱 회귀 (1) | 2024.07.17 |
